Projektübersicht
Die verschiedenen Projekte wurden konzipiert, um verlässliche Informationen zu den folgenden Themen zu liefern:
- In welchem Ausmaß sind resistente Enterobakterien in Mensch und Tier sowie in Lebensmitteln, Futtermitteln und der Umwelt verbreitet?
- Wo und welche Resistenzgene kommen in Enterobakterien vor (z.B. ESBLExtended Spektrum β-Lactamasen - und PMQRPlasmid-Mediated Quinolone Resistance-Gene)
- Welche neuen Resistenzgene treten auf?
- Wie sind die Übertragungswege zwischen Menschen, Tieren und der Umwelt?
- Was sind die Risikofaktoren für die Entstehung einer Infektion mit resistenten Enterobakterien beim Menschen?
- Was sind die Risikofaktoren für das Vorkommen resistenter Enterobakterien?
- Welches Risiko stellen resistente Enterobakterien, insbesondere von landwirtschaftlichen Nutztieren, tatsächlich für die menschliche Gesundheit dar?
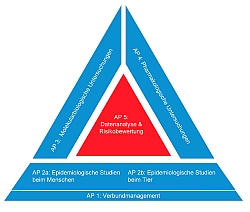
Die Projekte sind thematisch in fünf Arbeitsschwerpunkten zusammengefasst.
- Studien in Tieren, Lebensmittel und der Umwelt (AP 2a)
- Studien in der menschlichen Bevölkerung (AP 2b)
- Charakterisierung der Bakterien und der Resistenzeigenschaften (AP 3)
- Pharmakologische Experimente (AP 4)
- Risikobewertung (AP 5)
Die in den einzelnen Projekten erhobenen Daten werden in einer gemeinsamen Datenbank gespeichert und können so für die Projektübergreifende Analyse und Risikobewertung genutzt werden.
Bisher im Rahmen des Forschungsverbundes gewonnene Erkenntnisse:
- In einem hohen Anteil der untersuchten Betriebe mit Haltung von landwirtschaftlichen Nutztieren wurden Proben mit ESBL- bzw. AmpC produzierenden E. coli gefunden.
- Erstmalig wurden Carbapenemase-bildende E. coli und Salmonella bei landwirtschaftlichen Nutztieren in Deutschland gefunden (Fischer et al., 2012).
- Gemüsepflanzen (Weißkohl, Porree) sind in der Lage, aus Böden, die mit antimikrobiellen Rückständen belastetem Dünger beaufschlagt wurden, geringe Antibiotika in geringen konzentrationenKonzentrationen (Enrofloxacin) aufzunehmen.
- ESBL-E. coli können in und an Pflanzenkeimlingen gefunden werden
- Unter der Behandlung von Tiergruppen mit Antibiotika findet unabhängig von der Applikationsmethode eine Verschleppung in die direkte Umgebung der Tiere statt
- Auch die Exposition mit subtherapeutischen antimikrobiellen Konzentrationen übt einen Selektionsdruck aus und führt zu vermehrt auftretenden, in ihrer Sensibilität Empfindlichkeit reduzierten bis resistenten Bakterien (Scherz et al., 2014).
- Die ESBL-E. coli Kolonisationsrate bei gesunden Probanden liegt bei 6,3% (Valenza et al. 2013)
- Der Kontakt zu Hochendemiegebieten wie Asien ist ein Faktor, der mit einer Kolonisation mit ESBL-E.coli assoziiert ist (Leistner et al., 2013).
- Methoden zum Nachweis und zur Charakterisierung von Resistenzen vermittelt durch ESBL-Enzyme, PMQR und Carbapenemasen wurden harmonisiert und validiert.
- Eine "Next Generation Sequencing" (NGS) -Pipeline zur Plasmid- und Ganz-Genomsequenzierung wurde entwickelt. Dies ermöglicht eine schnellere Analyse und Lokalisation von Resistenzgenen sowie Pathogenitäts-assoziierten genetischen Strukturen (Virulenzgenen) in Plasmiden und im Bakteriengenom.
- Erstmalig wurden mittels Ganz-Genomsequenzierung ein deutschlandweit verbreiteter ESBL-Klon gefunden, der in Tier, Mensch, Umwelt und Lebensmittel zirkuliert (Falgenhauer et al. 2016).
- Die gemeinsame Datenbank ermöglicht die umfassende Erfassung und gemeinsame Analyse der in den unterschiedlichen Projekten erfassten Daten, zu Herkunft und Eigenschaften der Bakterienstämme.
- Modelle zur Quantifizierung des Beitrags von verschiedenen Quellen und zur Ausbreitung von ESBL-Bildner entlang der Geflügelschlachtkette wurden entwickelt.
- Auf Grundlage der gemeinsamen Stamm- und Datensammlung gelang, Anfang 2016 der schnelle Nachweis des übertragbaren Colistinresistenz (Mcr-1) in E. coli vom Nutztier (Falgenhauer et al. 2016).