Projektübersicht
Die verschiedenen Projekte wurden konzipiert, um verlässliche Informationen zu den folgenden Themen zu liefern:
- Wie sind die Erreger in Mensch und Tier sowie in Lebensmitteln, Futtermitteln und der Umwelt verteilt?
- Wo und welche ESBLExtended Spektrum β-Lactamasen - und PMQRPlasmid-Mediated Quinolone Resistance-Genen (Resistenzgenen) kommen in Enterobakterien vor?
- Welche neuen Resistenzgene treten auf?
- Wie sind die Übertragungswege zwischen Menschen, Tieren und der Umwelt?
- Was sind die Risikofaktoren für das Vorkommen resitenter Enterobakterien?
- Welches Risiko stellen resistente Enterobakterien, insbesondere von landwirtschaftlichen Nutztieren, tatsächlich für die menschliche Gesundheit dar?
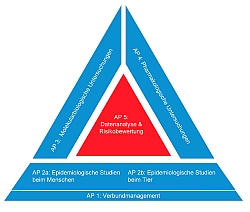
Die Projekte sind thematisch in fünf Arbeitsschwerpunkte zusammengefasst.
- Studien in Tieren, Lebensmittel und der Umwelt (AP 2a)
- Studien in der menschlichen Bevölkerung (AP 2b)
- Charakterisierung der Bakterien und der Resistenzeigenschaften (AP 3)
- Pharmakologische Experimente (AP 4)
- Risikobewertung (AP 5)
Von Tierkot, der Umgebung landwirtschaftlicher Betriebe und Lebensmitteln werden Proben entnommen. Es wird untersucht, wie oft Resistenzen in E. coli und S. enterica nachgewiesen werden. Zusätzlich wird der Einfluss verschiedener Risikofaktoren auf das Vorkommen der Erreger untersucht.
Die von in den einzelnen Projekten erhobenen Daten werden in einer gemeinsamen Datenbank gespeichert und können so für die Projektübergreifende Ananlyse und Risikobewertung genutzt werden.
Bisher im Rahmen des Forschungsverbundes gewonnene Erkenntnisse:
- Die gemeinsame Datenbank ermöglicht die umfassende Erfassung und gemeinsame Analyse der in den unterschiedlichen Projekten erfassten Daten, zu Herkunft und Eigenschaften der Bakterienstämme.
- In einem hohen Anteil der untersuchten Betriebe mit Haltung von landwirtschaftlichen Nutztieren wurden Proben mit ESBL-produzierenden Enterobakterien gefunden.
- Erstmalig wurden Carbapenemase-bildende Keime bei landwirtschaftlichen Nutztieren in Deutschland gefunden (Fischer et al., 2012).
- Wenn Pflanzen oder Tiere niedrigen Konzentrationen von Antibiotika ausgesetzt sind, können vermehrt resistente Bakterien gefunden werden.
- Der Anteil der ESBL-E. coli besiedelten Allgemeinbevölkerung: liegt bei ca. 6% (Valenza et al. 2013)
- Methoden zum Nachweis und zur Charakterisierung von Resistenzen gegen β-Laktam-Antibiotika mit erweitertem Wirkungsspektrum, (Fluor) Chinolonen und Carbapenemen wurden harmonisiert und validiert.
- Neuen Resistenzgenen und mobilen genetischen Elementen wurden identifiziert.
- Analyse und Lokalisation von Pathogenitäts-assoziierte genetische Strukturen (Virulenzgenen) im Bakteriengenom.
- Next Generation Sequencing (NGS) -Pipeline zur Plasmid- und Ganz-Genomsequenzierung wurde entwickelt.
- Modelle zur Quantifizierung des Beitrags von verschiedenen Quellen und zur Ausbreitung von ESBLs entlang der Geflügelschlachtkette wurden entwickelt.